Nat Biotechnol:研究人员开发新的机器学习模型预测CRISPR-Cas9编辑人原代T细胞的结果
来源:生物谷 2019-08-04 10:50
2019年8月4日讯 /生物谷BIOON /——在一项近日发表在《Nature Biotechnology》上、题为"Large dataset enables prediction of repair after CRISPR-Cas9 editing in primary T cells"研究中,来自Chan-Zuckerberg Biohub、斯坦福大学、加州大学等单位的研究人员在Alexa
2019年8月4日讯 /生物谷BIOON /——在一项近日发表在《Nature Biotechnology》上、题为"Large dataset enables prediction of repair after CRISPR-Cas9 editing in primary T cells"研究中,来自Chan-Zuckerberg Biohub、斯坦福大学、加州大学等单位的研究人员在Alexander Marson、Andrew P. May和James Zou的带领下,通过对基因编辑后的原代T细胞进行大规模测序获得一个数据库,并以此为基础建立了一种机器学习模型来预测原代T细胞在CRISPR-Cas9基因编辑发后的基因修复。
图片来源:Nature Biotechnology
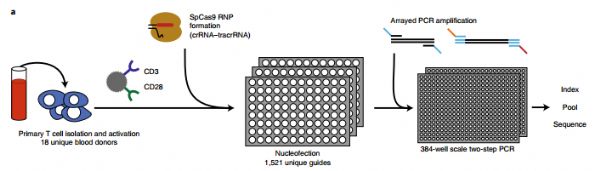
原发性T细胞是一种很有希望用于治疗性基因组编辑的细胞类型,因为它们可以在体外高效地进行工程设计,并可过继转移到患者身上。然而,目前还没有关于cas9依赖编辑的原代人类T细胞基因组编辑结果的详细信息。为此研究人员系统地描述了18例健康献血者原发性T细胞中的化脓性链球菌Cas9 (SpCas9)的修复结果。
研究人员对1656个人类原始T细胞靶基因位点的修复结果进行排序,并利用这些数据来训练一个机器学习模型,研究人员称之为CRISPR修复结果(CRISPR Repair Outcome,SPROUT)。
研究人员发现SPROUT可以准确预测核苷酸插入和删除的长度、概率和序列,这将有助于对具有重要治疗意义的原代T细胞的基因编辑过程中的SpCas9引导RNA的设计。(生物谷Bioon.com)
参考资料:
James Zou et al. Large dataset enables prediction of repair after CRISPR-Cas9 editing in primary T cells Nature Biotechnology. 2019. DOI https://doi.org/10.1038/s41587-019-0203-2
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->